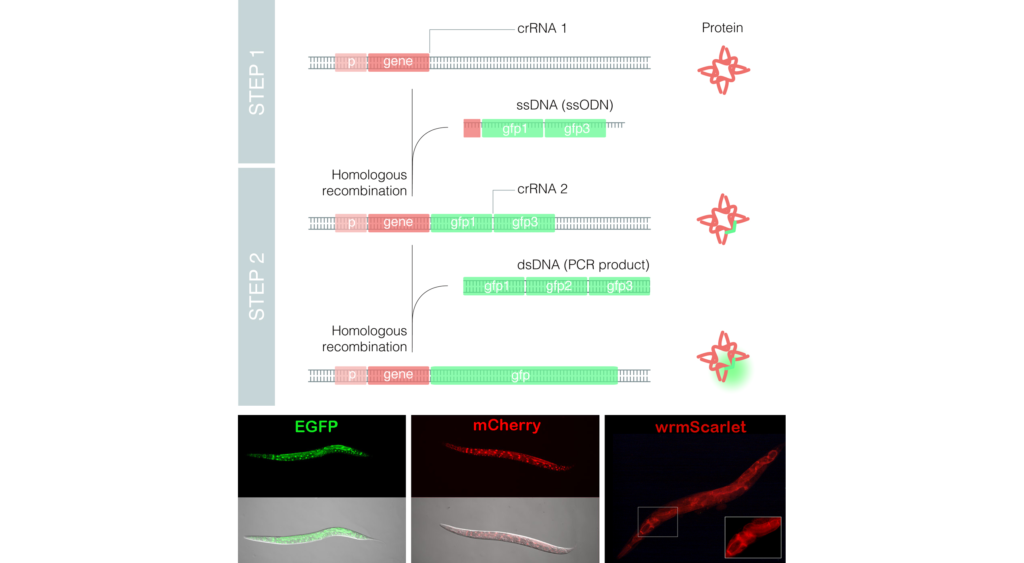
La técnica de CRISPR está revolucionando la investigación biomédica al permitir editar los genomas con gran precisión. Sin embargo, aunque CRISPR permite generar o corregir mutaciones de uno o de pocos nucleótidos con cierta facilidad, todavía presenta limitaciones a la hora de introducir en el genoma fragmentos de ADN más largos. Así, por ejemplo, la inserción en el genoma de un gen que produzca una proteína fluorescente como la GFP, de amplio uso en investigación, tiene una baja eficiencia y usa plásmidos que requieren clonajes complejos.
El grupo del Dr Cerón ha utilizado el animal modelo Caenorhabditis elegans para optimizar el uso de esta técnica y ha desarrollado el método Nested CRISPR. Este método, libre de clonajes, inserta fragmentos largos de ADN en dos pasos. En un primer paso introduce en el genoma una pequeña porción (menos de 200 nucleótidos) del fragmento largo, y esta pequeña secuencia sirve de “nido” o “plataforma de aterrizaje” para que el fragmento más largo (de una kilobase aproximadamente) se introduzca en el genoma en un segundo paso de una manera eficiente.
El trabajo, recientemente publicado en la revista Genetics, de la Sociedad Americana de Genética, está despertando un interés extraordinario alcanzando la máxima puntuación en los índices de repercusión de la revista. Animales modelo como C. elegans, con un ciclo de vida corto, permiten a los científicos investigar sobre las posibilidades y los límites del CRISPR. Este trabajo forma parte de la tesis doctoral de Jeremy Vicencio, que tiene la prestigiosa beca INPhINIT de La Caixa. Tanto Jeremy como las investigadoras predoctorales Carmen Martínez y Xènia Serrat han realizado cientos de microinyecciones en la línea germinal de C. elegans y miles de genotipados para demostrar de una manera sólida y convincente la eficiencia de la nueva técnica Nested CRISPR.
Como moléculas reparadoras de ADN, Nested CRISPR usa oligonucleótidos comerciales para el primer paso y productos de PCR universales para el segundo. Esto facilita experimentos a gran escala que podrían etiquetar cientos de genes con proteínas fluorescentes de interés. Nested CRISPR también es modular y a esas proteínas fluorescentes se les pueden añadir otros péptidos u otras proteínas de interés. Así, por ejemplo, el grupo está trabajando en el marcaje de estas proteínas fluorescentes con péptidos que permiten su degradación de un modo controlado por el investigador.
Por último, el grupo de modelos de enfermedad en C. elegans que dirige el Dr Cerón pretende usar Nested CRIPSR para reemplazar genes de C. elegans con genes humanos. De este modo, este pequeño nematodo podría servir de modelo multicelular para estudiar el efecto de mutaciones humanas relacionadas con enfermedades. Este sistema tendría bastante interés en el campo de la medicina personalizada ya que de una manera rápida y eficiente se podría tener un pronóstico sobre la patogenicidad de una mutación o de una variación del genoma (conocidas como polimorfismos).